Hintergrundinformation
Das Leben
Vererbung, Gene & DNS
Dass bei der Vermehrung von Lebewesen Eigenschaften an die Nachkommen weitergegeben werden, war angesichts der Ähnlichkeit von Kindern mit ihren Eltern schon in der Antike klar. Über die Ursachen gab es viele Spekulationen: so wurde vermutet, dass über den ganzen Körper verteilt Samenzellen entstanden, die dann über das Blut in den Penis transportiert wurden und bei der Fortpflanzung die Eigenschaften “ihrer” Organe weitergaben. So konnten auch erworbene Eigenschaften weitergegeben werden: Durch Arbeit gestärkte Muskeln etwa ergaben stärkere Samenzellen. Erst um 1885 stellte der Freiburger Zoologe August Weismann die Theorie auf, dass es im Körper eigene Keimzellen gäbe, die die Erbinformationen enthielten, die nicht durch Veränderungen im Körper verändert würden. Denkbarer Träger waren für ihn die Kernkörperchen der Keimzellen, die auf die Nachkommen übertragen würden.
Wichtige Grundlagen der Vererbung hatte – seinerzeit fast unbemerkt – schon Mitte des 19. Jahrhunderts der Augustinermönch und Naturforscher Gregor Mendel in einem Klostergarten in Brünn (heute Brno, Tschechische Republik) entdeckt. Mendel, der zuvor in Wien Naturwissenschaften studiert hatte, aber bei der Lehramtsprüfung durchgefallen war, fand unter anderem durch sorgfältig angelegte Versuche an Gartenerbsen heraus, dass Merkmale der Erbsen dominant (vorherrschend) oder rezessiv (verborgen) sein können. Kreuzt man etwa glatte Erbsen mit schrumpeligen Erbsen, sind alle Nachkommen glatt. Bei anderen Merkmalen können die die Nachkommen “intermediär” sein, also zwischen den Eltern stehen – rot- und weißblühende Eltern ergeben z.B. rosablühende Nachkommen. Unabhängig davon, ob die Vererbung eines Merkmals dominant/rezessiv oder intermediär erfolgt, sind die Nachkommen untereinander immer gleich. (Dies ist die 1. Mendelsche Regel – oder “Uniformitätsregel”). Kreuzt man aber die Nachkommen der glatten und schrumpeligen Erbsen – die alle glatt sind – untereinander, entwickeln sich runde und schrumpelige Erbsen im Verhältnis 3:1 (2. Mendelsche Regel – oder “Spaltungsregel”). Mendel erklärte dies damit, dass Merkmale von “Faktoren” (heute Gene genannt) übertragen werden, die in verschiedenen Formen (heute Allele genannt) vorliegen können, von denen eine dominant ist (das erkennbare Merkmal bestimmt), der andere verborgen (“rezessiv”). Die Faktoren treten in Paaren auf, bei der Fortpflanzung gibt aber jedes Elternteil nur eine Form an die Nachkommen weiter, dies erklärt die “Aufspaltung” des Merkmals in der 2. Generation (siehe Abbildung).
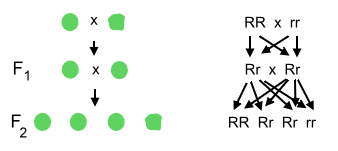
Mendels Kreuzungsversuch mit
runden und schrumpeligen Erbsen: In der 1. Generation
(F1)
sind alle Nachkommen untereinander gleich, in der zweiten (F2)
spalten sie sich wieder auf. Eigene Abbildung.
Mendel fand auch heraus, dass verschiedene Merkmale (etwa Form der Samen und Blütenfarbe) sich unabhängig voneinander vererbten, Faktoren (Gene) also unabhängig voneinander weitergegeben werden (3. Mendelsche Regel – “Unabhängigkeitsregel”). Mendels Ergebnisse wurden um 1900 mehrfach (jeweils unabhängig voneinander) wiederentdeckt, und gingen eine fruchtbare Verbindung mit neuen Ergebnissen der Zellbiologie ein. 1879 hatte der deutsche Biologe Walther Flemming eine färbbare Substanz im Zellkern entdeckt und “Chromatin” genannt, 1888 prägte der Anatom Heinrich Wilhelm Waldeyer das Wort “Chromosom” für die während der Zellteilung erkennbaren Kernkörperchen (wie sich herausstellen sollte, sind beide das gleiche, Chromosomen sind während der Zellteilung verdichtetes Chromatin, siehe >> unten). 1902 zeigten der amerikanische Biologe Walter S. Sutton und sein deutscher Kollege Theodor Boveri basierend auf Weismanns Idee von den Kernkörperchen als Träger der Erbinformation, dass Chromosomen sich genau wie Mendels Erbfaktoren verhielten (306); so entstand die Chromosomentheorie der Vererbung. 1910 konnte Thomas Hunt Morgan durch Kreuzungsversuche mit der Taufliege Drosophila melanogaster zeigen, dass die Chromosomen tatsächlich die Träger der Erbinformation sein mussten: Er erklärte ein von den Mendelschen Regel abweichendes Ergebnis von Kreuzungen weiß- und rotäugiger Taufliegen damit, dass das betreffende Gen auf dem X-Geschlechtschromosom lag – die Geschlechtschromosomen sind die einzigen, die nicht als Paar vorkommen, sondern beim Mann als unterschiedliche Chromosomen (meist XY benannt) – für Gene, die auf dem X-Geschlechtschromosom liegen, haben männliche Organismen daher oft kein Gegenstück (manchmal doch, dann liegen diese auf dem Y-Chromosom). Hunt Morgan bekam für diese Entdeckung 1933 den Nobelpreis für Medizin. Damit stellte sich den Biologen eine neue Frage: Wie können die winzigen Chromosomen im Zellkern die Merkmale ganzer Organismen bestimmen?
Die Entdeckung der DNS als Erbsubstanz
Diese Frage ließ sich leichter an Viren und Bakterien untersuchen, deren Erbmaterial viel einfacher strukturiert ist als bei Eukaryoten mit Zellkern. 1943 entdeckten der kanadische Mediziner Oswald Avery und seine Arbeitsgruppe, dass Pneumokokken (die Erreger der Lungenentzündung) von der harmlosen R-Form in die krankheitserregende S-Form übergehen, wenn man sie mit abgestorbenen Zellen der S-Form mischt. Diese Veränderung ging auf die Nachkommen über, war also erblich. Avery nahm an, dass die Gene also als als chemische Substanz in den abgestorbenen Zellen enthalten sein mussten. Wie sich herausstellte, war es eine 1869 von dem deutschen Mediziner Friedrich Miescher in den Zellkernen entdeckte Substanz, die Desoxyribonukleinsäure (DNS – auch bekannt unter der englischen Abkürzung DNA), mit der die Eigenschaften von der S-Form auf die R-Form übertragen wurden: Diese DNS – es war bereits bekannt, dass sie in den Chromosomen vorkam – musste also der Träger der Erbinformation sein. Bestätigt wurde Averys Entdeckung 1952, als Alfred Hershey und Martha Chase zeigten, dass ein Virus, der das Bakterium Escherichia coli befällt, nicht selbst in die Bakterienzelle eindringt, sondern von außen DNS in die Zelle einschleust – und diese dann neue Viren herstellt. Daher muss die DNS die Information enthalten, die für die Vermehrung der Viren erforderlich ist (die DNS “kapert”, wie wir heute wissen, mit dem Einbringen ihrer Erbinformation den biochemischen Apparat der Zelle).
(Der Mensch hat in seinen Zellen 46 Chromosomen. Würde man die in einer Zelle enthaltene DNS geradeziehen und aneinanderlegen, ergäbe sich eine Länge von über zwei Metern – in jeder Zelle, die ja nur einen Durchmesser von einigen Tausendstel Millimetern hat. Alle DNS-Abschnitte im menschlichen Körper zusammen ergäben geradegezogen und aneinandergelegt eine Länge von 20 Milliarden Kilometern – oder 65-mal zur Sonne und zurück! [316])
Das Verständnis der molekularen Funktion
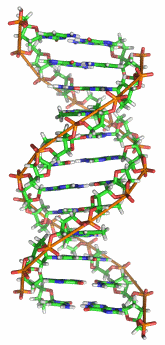
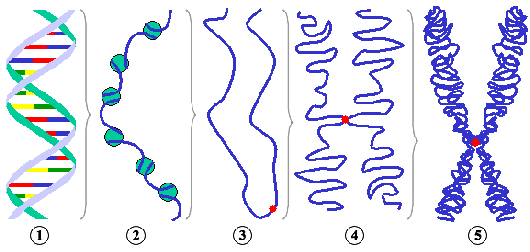
DNS-Doppelhelix. Abb. von Richard Wheeler aus
>>
wikipedia, abgerufen 2.11.2008. Lizenz; >> FDL
Mit diesen Erkenntnissen war die DNS als Erbsubstanz entdeckt. Damit ergaben sich neue Fragen: Wie konnte sich die DNS verdoppeln, so dass sei bei der Zellteilung an alle Tochterzellen weitergegeben wird? Und wie wird in der DNS Information verschlüsselt? Die Bestandteile der DNS – Basen, Zucker und Phosphatreste – waren schon seit 1929 bekannt, und 1953 entdeckten der Amerikaner James Watson und der Engländer Francis Crick (anhand von Röntgenbeugungsbildern, die die englische Biochemiker Rosalind Franklin und ihre Kollegen Raymond Gosling und Maurice Wilkins aufgenommen hatten) die dreidimensionale Struktur der DNS: die berühmte Doppelhelix (Abb. rechts). Die Zucker und Phosphatreste bilden wie die Holme einer verdrehten Leiter zwei gewundene, umeinander geschlungene Spiralen, verbunden sind diese durch Wasserstoffbrückenbindungen der im Inneren der Doppelhelix liegenden Basen; diese bilden gewissermaßen die Sprossen der verdrehten Leiter.
Schon 1950 hatte Erwin Chargaff gefunden, dass von den vier in der DNS vorkommenden Basen – Adenin (A), Cytosin (C), Guanin (G) und Thymin (T) – A und T sowie C und G jeweils in den gleichen Mengen vorkamen. Watson und Cricks zeigten, warum: In der Doppelhelix konnten sich nur A und T sowie C und G paaren, die Sprossen bestehen also nur aus den Basenpaaren A-T oder C-G. Diese Erkenntnis legte sofort nahe, wie sich die DNS verdoppeln konnte: Trennt man den Doppelstrang auf, bilden die am Holm verbleibenden Basen eine Matrize: da sie sich immer nur mit dem gleichen Base paaren (A mit T, T mit A, C mit G, G mit C), können aus zwei Elternsträngen nach Bildung der entsprechenden Tochterstränge zwei identische DNS-Moleküle werden. (Dass dieses so ist, zeigten Matthew Meselson und Franklin Stahl 1958 mit schweren Stickstoff-Isotopen: Neue DNS entsteht immer nur an einer Seite der Doppelhelix.)
Wie diese Verdoppelung funktioniert, entdeckten 1956 Arthur Kornberg und sein Team: Sie isolierten ein Enzym, die DNS-Polymerase, die für die schrittweise Zufügung neuer Bausteine an eine DNS-Kette sorgt (1959 bekam Kornberg hierfür den Nobelpreis). Zuvor wird die Doppelhelix durch andere Enzyme, die Topoisomerase und die Helikase (-ase ist eine Endung, die für ein Enzym steht), entspiralisiert und aufgetrennt, und dann werden an die so entstandenen Einzelstränge durch DNS-Polymerasen die Gegenstränge angelagert, beginnend an einem RNS-Primer genannten “Starter”. Dabei baut jede DNS-Polymerase nur Fragmente, die von einer Ligase zusammengefügt werden.
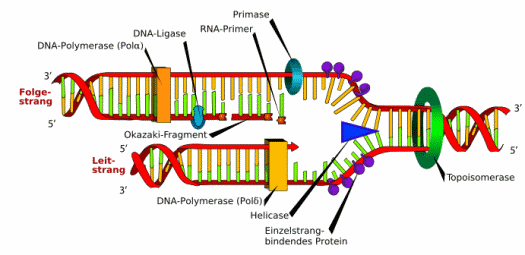
Verdoppelung (“Replikation”) der DNS-Doppelhelix. Abb.: Mariana Ruis Villarreal, aus >> wikipedia, public domain, abgerufen 2.11.2008.
(Eine Methode, Art und Reihenfolge (Sequenz) der Basen in der DNS zu bestimmen, entwickelte in den 1970er Jahren der britische Biochemiker Fred (eigentlich Frederick) Sanger, der hierfür 1980 zum zweiten Mal den Chemie-Nobelpreis erhielt [den ersten hatte er 1958 für seine Arbeiten zur Sequenzierung von Proteinen und zur Aufklärung der Struktur von Insulin erhalten].)
Von der DNS zum Protein
Die Frage, wie die DNS Information verschlüsselte, erkannten im Jahr 1961 der deutsche Biochemiker Heinrich Matthaei und sein amerikanischer Kollege Marshall Warren Nirenberg: sie "knackten" den genetischen Code und fanden heraus, dass die Basen (bestimmter Abschnitte) der DNS auch die Informationsträger des Moleküls sind – sie enthalten Informationen, die die Zellen zum Aufbau von Proteinen nutzen. Je drei Basen codieren eine Aminosäure. Das Alphabet der DNS besteht also aus 4 Buchstaben (den Basen A, C, G und T), die Worte aus drei Basen bilden, zum Beispiel GCT, CGA oder TTC. Ein solches “Wort” heißt in der Genetik Codon, und jedes Codon entspricht einer Aminosäure im Protein. (GCT steht zum Beispiel für die Aminosäure Alanin. Von den 64 möglichen Kombinationen werden nur 20 Aminosäuren codiert, das heißt, mehrere ”Worte” codieren die gleiche Aminosäure.) Bei der Übersetzung Basen – Aminosäure spielt die Ribonukleinsäure (RNS) eine entscheidende Rolle. RNS ist ein der DNS sehr ähnliches Molekül. Im ersten Schritt, der Transkription, entsteht durch ein RNS-Polymerase genanntes Enzym eine messenger-RNS (m-RNS, “Boten-RNS”). Sie ist der Botenstoff, der die Information der DNS zu den Ribosomen transportiert, kleinen Körperchen im Zellinneren, an denen der Aufbau von Proteinen stattfindet. Die m-RNS enthält genau die gleiche Information wie der DNS-Abschnitt, aus dem sie hergestellt wurde. An der DNS entstehen weitere RNS-Moleküle: Die transfer-RNS (t-RNS) und die ribosomale RNS (r-RNS). Die r-RNS bildet gemeinsam mit mehreren Dutzend Proteinen die Ribosomen; die t-RNS vermittelt zu jedem Codon die passende Aminosäure: Sie besitzt zum einen einen Anticodon; der aus den drei Basen besteht, die zur Paarung mit dem Codon geeignet sind – so lagert sich immer die “richtige” t-RNS an die m-RNS an – und eine spezifische Aminosäurebindungsstelle, an der die entsprechende Aminosäure hängt. So bringt ein t-RNS-Molekül nach dem anderen jeweils die nächste, vom Codon codierte Aminosäure zum Ribosomen, wo diese zu Aminosäureketten verknüpft werden – den Proteinen (diesen Schritt nennen die Biologen Translation, “Übersetzung”).
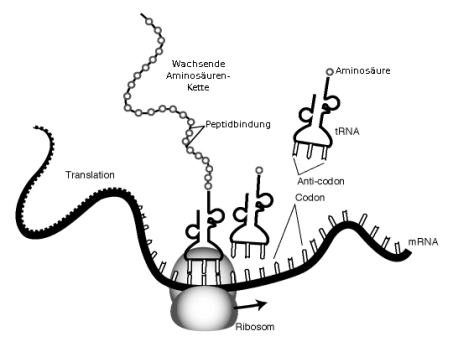
Ablauf der Proteinbiosynthese.
An den Ribosomen treffen m-RNS und t-RNS zusammen, die von der t-RNS
mitgeführten Aminosäuren werden zu Proteinen verknüpft. Abb.: www.genome.gov,
public domain.
Nur ein Teil der DNS besteht aus Bereichen, die tatsächlich zu Proteinen “übersetzt” werden – diese heißen Exons. Der andere, je nach Art unterschiedlich große Teil der DNS besteht aus Introns, nicht codierenden DNS-Anteilen. Diese bestehen zumindest teilweise offenbar aus “altem Code”, der im Laufe der Stammesgeschichte nicht mehr gebraucht wurde. Zumindest bei Hefen kann dieser in Stresszeiten aber noch aktiviert werden; und funktionslos sind die Introns sowieso nicht: Zum einen kodieren sie zwar keine Gene, aber RNS-Abschnitte, die bei der Genregulation eine Rolle spielen (siehe unten), zum anderen bestimmen sie mit ihren Basensequenzen die räumliche Faltung und die Möglichkeiten zur Bindung an Proteine und beeinflussen daher die Form und Ablesbarkeit des DNS-Doppelstranges. Die Einheiten der Erbinformation werden als Gen bezeichnet. Bei den Eukaryoten kann sich ein Gen auch aus mehreren Exons zusammensetzen. Die Introns werden in diesem Fall vor der Proteinbiosynthese noch im Zellkern aus der m-RNS herausgeschnitten; dieser Vorgang wird splicing oder “Spleißen” genannt.
Zu einem Gen gehören auch Sequenzen, die die Ablesung der entsprechenden DNS regulieren (regulatorische Sequenzen); denn ob ein Gen in ein Protein übersetzt wird, wird von vielen Faktoren entscheiden, die über diese regulatorischen Sequenzen wirken – hier können Gene ab- und angeschaltet werden. Dies ist auch eine Form, wie Gene auf ihre Umwelt reagieren können: Wenn sich Methylgruppen an bestimmte Abschnitte heften, können Gene abgeschaltet werden (diesen Einfluss der Umwelt auf die Gene untersucht die Epigenetik, ein aktuelles Feld der genetischen Forschung). Gene können sich auch verändern, diese Veränderungen nannte ihr Entdecker, der niederländische Biologe Hugo de Vries, Mutationen. Mutationen entstehen durch äußere Einwirkungen auf die DNS (z.B. UV-Strahlung oder bestimmte chemische Stoffe) sowie durch Fehler bei der DNS-Verdoppelung Eine solche Mutation kann die Veränderung einer einzigen Base in einem Exon sein, die einem Gen einen ganz neuen Sinn geben kann (wie aus “Hans geht fort” “Hans weht fort” wird).
(Mutationen sind sehr selten, auch weil sehr wirksame Mechanismen in den Zellen "Fehler" in der DNS aufspüren. Man schätzt, dass bei rund einer Milliarde kopierter Basen ein "Fehler" überbleibt. Diese geringe Rate ist Grundlage der großen Stabilität von Erbanlagen - unsere DNS ist z.B. immer noch zu über 99 Prozent mit der von "Frühmenschen" identisch. Aber sie ist auch groß genug, um die Variabilität zu ermöglichen, an der die Evolution ansetzt, die über extrem lange Zeiträume die heutige Vielfalt des Lebens ermöglicht hat.)
Die Erbsubstanz der meisten Säugetiere besteht aus etwa 30.000 Genen; die Zahl der Gene unterscheidet sich zwischen den Arten nicht sehr. Der englische Evolutionsbiologe Richard Dawkins hat die Gene mit Subroutinen eines Computerprogramms verglichen: Diese sind in vielen Programmen gleich, der Unterschied in den Programmen kommt durch die Reihenfolge der Aktivierung der Subroutinen zustande. Ganz ähnlich bestimmt die Reihenfolge und die Kombination der Aktivierung der Gene die wichtigsten Unterschiede zwischen biologischen Arten – oder auch innerhalb der Generationen einer Art und sogar zwischen verschiedenen Gewebezellen: So besitzen ja Raupen und Schmetterlinge oder Knochen- und Leberzellen das gleiche Genom; der Unterschied in der Erscheinung wird nur durch die unterschiedliche Genaktivierung ausgelöst. Wie genau diese Aktivierung gesteuert wird, ist eines der aktuellsten Forschungsgebiete der Genetik, wobei sich die Erbsubstanz als komplizierter erweist, als früher angenommen: Da gibt es “springende Gene”, die ihre Position auf den Chromosomen verändern können; Gene, die (durch unterschiedliche Verarbeitung von Vorstufen der m-RNS – “alternatives Spleißen”) mehrere Proteine kodieren können; Introns, die zu großen Teilen in RNS-Abschnitte “übersetzt” werden, die Gene ganz oder teilweise abschalten können (“RNS-Interferenz”) – die Vererbung besteht aus einem komplexen, raumzeitlichen Zusammenspiel von DNS, RNS und Proteinen (und, wie oben dargestellt, Methylgruppen), das noch niemand richtig verstanden hat.
Im Prinzip verläuft die Entwicklung wohl so: Wenn bei der Befruchtung das Spermium an die Eizelle andockt, gehen von dieser Stelle elektrische Impulse aus, die die Bildung von Botenstoffen auslösen, deren Konzentration festlegt, wo vorne und hinten, unten und oben ist. Diese Konzentrationsunterschiede werden bei den ersten Zellteilungen auf die Tochterzellen übertragen, und wirken dort auf Steuerungsgene, deren Aktivierung den Aufbau komplexer Strukturen (etwa den Flügel einer Fliege oder den Arm eines Menschen) auslöst. Diese Steuerungsgene heißen Homöobox-Gene; sie “kennen” ihre Position im Körper durch die Konzentration bestimmter Stoffe und lösen in ihren Zellen durch die Produktion von Eiweißmolekülen, die wiederum auf andere Gene wirken, die Bildung der “richtigen” Strukturen und Organe aus. Diese Homöobox-Gene sind bei allen Tieren miteinander verwandt: Fliegen besitzen acht dieser Gene, Säugetiere 28 – ausschließlich abgewandelte Fliegen-Homöobox-Gene, sie sind also wahrscheinlich durch Verdoppelung von Fliegengenen entstanden. Sogar bei Seeanemonen hat man diese Gene inzwischen entdeckt; dieser universale Mechanismus wurde offenbar über die ganze Evolution im Prinzip beibehalten. Die sich entwickelnden Zellen tauschen auch Stoffe mit ihren Nachbarzellen aus, wodurch eine Art Feinkoordination sichergestellt wird, bis alle Organe des Körpers richtig ausgebildet sind. Drei Prozesse wirken also zusammen: Zellteilung, Zelldifferenzierung (die Ausbildung spezialisierter Zellen)und die Gestaltbildung (das Zusammenlagern von Zellen zu Geweben und Organen). Hierauf folgt das Größenwachstum, und dabei müssen die Zellen “wissen”, wann sie aufhören müssen, sich zu teilen.
Das Ergebnis nennen die Biologen “Phänotyp” – die Summe aller erkennbaren Merkmale eines Lebewesens. Dabei sind nicht nur äußerliche Merkmale gemeint, sondern z.B. auch biochemische Merkmale. “Erkennbar” sind die Merkmale auch (und sogar vor allem) für die Evolution: Die “natürliche Auslese” wirkt auf die Phänotypen; dieser ist das Ergebnis, anhand derer die Gene von der Evolution “beurteilt” werden – genau genommen nicht Gene, sondern wie oben beschrieben Gen-Kooperationen: Wenn diese zu einem besser angepassten Phänotyp führen, werden sie bevorzugt in die nächste Generation weitergegeben. Weiter kompliziert wird die Wirkung der natürlichen Auslese auf die Gene dadurch, dass sich ein Gen oft (meist?) auf mehr als ein Merkmal auswirkt; eine (im Sinne der Evolution) negative Auswirkung auf ein Merkmal also durch eine positive Auswirkung auf ein anderes Merkmal ausgeglichen werden kann.